Tarkka-geenin poistaminen ja korvaaminen käyttäen CRISPR/Cas9-järjestelmä ihmisen soluissa
Tässä osoitamme, että kahden opas RNAs yhdistettynä Cas9 tehokkaasti tuottaa DNA-poistot enintään 10 kt ihmisen soluissa on prosessi, jossa korjaus poisto on pitkälti suoritettu by tarkka end liittyä. Lisäksi toimitamme tietoja, jotka osoittavat, että CRISPR / Cas9-järjestelmä voi korvata suuria genomisia fragmentteja lineaarisen homologisen korjausluovuttajan läsnä ollessa.,
bakteeri aihekokonaisuuksien säännöllisesti ryhmittyneinä lyhyt palindrominen toistaa/ CRISPR—liittyvät (CRISPR/Cas) loci koodaa RNA-ohjattu immuunijärjestelmä, joka suojaa soluja vastaan hyökkääviä viruksia ja plasmidit (1, 2). Streptococcus pyogenes, tyyppi II CRISPR/Cas-järjestelmät käyttävät RNA-ohjattu endonuclease (RGEN), Cas9, katalysoida laitoskohtaiset pilkkominen kohde-DNA-sekvenssit., Kohdistaminen Cas9 erityisiä genomista sivustoja välittyy 20 nukleotidin opas sekvenssin sisällä liittyvä CRISPR RNA (crRNA) ja vaatii trans-aktivointi crRNA (tracrRNA), joka rekrytoi crRNA osaksi Cas9 monimutkainen (3). Tunnustaminen pilkkominen sivustoja määräytyy crRNA-DNA base pariliitoksen ja protospacer vieressä motiivi (PAM), kolmen nukleotidin sekvenssi (NGG) rinnakkain DNA täydentäviä alueella (4)., On huomattava, että yksi opas-RNA (gRNA), joka jäljittelee tracrRNA-crRNA monimutkainen voi rekrytoida Cas9 kohdennettuja genomista sivustoja ja tuottaa double-stranded taukoja (DSBs) DNA (5). CRISPR / Cas9-järjestelmiä on mukautettu paikkakohtaiseen genomin muokkaukseen erilaisissa solutyypeissä ja-organismeissa (6-12).
Genomin editointi CRISPR/Cas9 on aloitettu ottamalla käyttöön DSB: n on kohdennettu genomisen lokuksen käyttäen RNA-ohjelmoitu RGEN. Tämän jälkeen korjaus DSB: n kautta joko homology-suunnattu korjaus (HDR) tai nonhomologous end-liittymällä (NHEJ)., Läsnä homologisia korjaus luovuttajan, CRISPR/Cas9-järjestelmää voidaan käyttää tuottamaan tarkat ja määritellyt muutokset ja lisäykset on kohdennettu locus läpi HDR prosessi. Ilman homologisia korjaus luovuttajan, yhden DSBs syntyy CRISPR/Cas9 on korjattu läpi virhe-altis NHEJ, joka johtaa lisäys tai poisto (indel) mutaatioita. Indel mutaatioita koodaus eksonien voivat ottaa käyttöön ennenaikainen stop kodonien tai frame-shift-mutaatiosta, mikä inaktivoi vastaavat proteiinit., Indel mutaatiot syntyvät korjaus yhden DSB voi olla hyödyllinen kokeita, joilla pyritään kuvaavat toiminnallinen verkkotunnukset proteiinia koodaavan geenien tai inaktivointi genomista elementtejä, kuten intergenic tai intronic sääntely-sekvenssit tai noncoding RNA-geenit. DNA-fragmentti poistot kohde loci antaisi avenue tutkia näitä toiminnallisia elementtejä. Tämän vuoksi, useita DSBs on otettu käyttöön tuottaa poistot Drosophila (12, 13), seeprakala (14), ja ihmisen soluja (8), vaikkakin alhainen tehokkuus., Kohdennettuja genomisia DNA-poistoja on saavutettu myös käyttämällä sinkkisormen nukleaasia (ZFN) tai transkription aktivaattorin kaltaista efektorinukleaasia (TALEN) ihmissoluissa (15-17). Näiden lähestymistapojen tehokkuus on kuitenkin yleensä vähäistä. Lisäksi ZFNs ja TALENs edelleen hieman vaikeaa ja kallista suunnitella, kehittää ja empiirisesti testata matkapuhelinverkon yhteydessä.
Tässä tutkimme sukupolven fragmentti poistot ihmisen soluissa katalysoivat CRISPR/Cas9-järjestelmä. Osoitamme, että 2 gRNAs yhdistettynä Cas9 voi tehokkaasti luoda DNA poistoja jopa 10 kb., Olemme huomanneet, että tämän poistoprosessin korjaaminen tapahtuu suurelta osin täsmällisellä liittymisellä. Lisäksi kohdennettu poistaminen CRISPR/Cas9-järjestelmällä näyttää olevan riippumaton kohdennetun lokuksen transcriptionaalisesta asemasta. Lopuksi osoitamme, että CRISPR/ Cas9-järjestelmä voidaan korvata suuri genomista palasia läsnä lineaarinen homologisia korjaus luovuttajan.
aineisto ja Menetelmät
Plasmidi rakentaminen
perus H1 promoottori oli monistettu pLVTHM plasmidi (Addgene, #12247, Cambridge, MA)., Oligonukleotidit, jotka sisälsivät muunneltua H1-promoottoria ja haluttujen gRNA-sekvenssien selkärankaa kahdella BsaI-alueella, syntetisoitiin (Pan Facility, Stanfordin yliopisto). Tuloksena olevat täyspitkät tuotteet vahvistettiin PCR: llä ja kloonattiin pUC19-vektoriksi. Ampisilliini-geenin (amp) ja H1 järjestäjä pUC19 vektori sisältää BsaI restriction entsyymi sivustoja; nämä olivat mutatoitunut (amp-geeni oli muuttunut G1601C, joka ei muuta aminohappo järjestyksessä; H1 promoottori oli muuttunut GAGACC että GAGGACC) poistamaan BsaI-sivustot., Grna-kloonausta koskeva pöytäkirja esitetään Lisäaineistossa. Kaikki kohdesarjat esitetään täydentävässä taulukossa S1.
Cell culture
HEK 293T, SK-Hep1, ja HeLa-solut viljeltiin vuonna Dulbecco”s modified Eagle”s medium (DMEM) täydennettynä 10% naudan sikiön seerumia (FBS) (Hyclone, Logan, UT) ja penisilliini/streptomysiini (pen/strep) (Invitrogen, Carlsbad, CA). PC3-soluja viljeltiin rpmi-1640-väliaineessa, jota täydennettiin 10% FBS: llä ja pen/strep: llä., Esimerkiksi tuumorinekroositekijä α (TNF-α) stimulaatio, 293T-soluja hoidettiin ilmoitettu pitoisuudet TNF-α(R&D Systems, Minneapolis, MN). Solut säilyivät 37°C: ssa ja 5% CO2: ssa kostutetussa hautomossa.
DNA poistaminen
HEK 293T-solut istutettiin 12-kuoppalevyillä, kun tiheys on 100 000 solua per kaivo. Jälkeen 24 h, solut olivat hetkellisesti transfektoiduissa 1 µg Cas9 plasmidi (Addgene, #41815), 0,5 µg gRNA T1, ja 0,5 µg gRNA T2 plasmidit käyttäen Lipofectamine 2000 (Invitrogen) kohti valmistajan”s protokollia., Genomien DNA uutettiin 48 tuntia transfektion jälkeen käyttäen QuickExtract DNA Extraction Solution (Biotekniikan Keskus, Madison, WI). Yhteinen PCR-järjestelmä toteutettiin kohdealueen vahvistamiseksi käyttämällä kohdealueita täydentäviä pohjamaaleja. Villityyppiset ja typistetyt genomifragmentit ratkaistiin geelielektroforeesilla. Reaaliaikainen PCR (RT-PCR) suoritettiin kvantifioida prosenttiosuus deleetio käyttäen primers poikki risteyksen tai sisällä poisto alueella. Vertaileva Cq menetelmää käytettiin laskea ilmaisun tason tavoite alueen suhteellinen viittaus alueen (ACTB locus)., Prosentuaalinen poistuma Kohdealueella laskettiin edelleen kohdesolujen suhteella kontrollisoluihin. Kaikki primer-sekvenssit on lueteltu täydentävässä taulukossa S2.
Tavoite sekvensointi
Solut kerättiin kaksi päivää transfektion jälkeen, ja genomien DNA uutettiin käyttämällä QuickExtract DNA Extraction Solution (Biotekniikan Keskus). PCR suoritettiin täydentää kohdistaminen alueen genomista DNA: ta on johdettu solut, ja amplicons oli syvä järjestyksessä MiSeq Henkilökohtainen Sekvensseri (Illumina, San Diego, CA).,
Targeted DNA replacement
The linear donor was generated by PCR from pGl3-GFP-SV40pA plasmid, created by replacing the Renilla gene with the GFP gene in pRL-TK (Promega, Madison, WI). The primer sequences used for PCR were:
CCL2-donor-F
A*C*AGCAGCCAGAGGAACCGAGAGGCTGAGACTAACCCAGAAACATCCAATGCTTTTACGCGTCCTAGCG
CCL2-donor-R
C*A*AAAATATATTTATTTGGTGTAATAG TTACAAAATATTCATTTCCACAACCACCTGGATCCTTATCGA
The underlined regions indicate the termini of analogous oligonucleotides with 50 bp of CCL2 homology., Luovuttajasekvenssit esitetään Lisäaineistossa. Kaksi 5′-yhteyttä ovat fosforotioaattia (merkitty tähdellä). Solujen 6-kuoppalevyillä oli hetkellisesti transfektoiduissa 2.0 µg Cas9 plasmidi, 0,8 µg gRNA T1 plasmidi, 0,8 µg gRNA T2 plasmidi, ja 0,4 µg lineaarinen luovuttajan käyttäen Lipofectamine 2000 (Invitrogen). 48 tunnin kuluttua siirrosta soluja hoidettiin 1 ng/mL TNF-α: lla 24 tunnin ajan, minkä jälkeen GFP-positiiviset solut lajiteltiin.
Luciferase assay
Varten luciferase assay, HEK 293T-solut istutettiin 96-kuoppalevyillä, kun tiheys on 5000 solua per kaivo., Jälkeen 24 h, solut olivat hetkellisesti transfektoiduissa 5 ng pRL-TK Renilla luciferase toimittaja ja 100 ng luciferase toimittaja sytomegalovirus (CMV), SV40 (Simian virus 40), tai perus-promoottori. 48 tunnin kuluttua luciferaasin aktiivisuutta mitattiin dual luciferase reporter assay-menetelmällä (Promega).
Western blot
Proteiinit erotettiin sodium dodecyl sulfate—SIVU (SDS-PAGE) ja siirretään nitroselluloosa kalvoja. Kalvot olivat tukossa 5% rasvatonta maitoa ja inkuboitiin kanssa GFP-vasta-aine (CST, #2555S, Danvers, MA)., Antigeeni-vasta-ainekompleksi havaittiin tehostetuilla kemiluminesenssireagensseilla.
Tulokset ja keskustelu
Me mukautettu bakteerien tyypin II CRISPR/ Cas9-järjestelmän mutagenize genomista DNA: ta ihmisen soluja. Ihmisen kodonissa-optimoitu versio S. pyogenes-Cas9 proteiinia, joissa on C-terminaalissa SV40 nuclear localization signal oli ilmaistu käyttäen aiemmin kuvattu järjestelmä (6). Suoraan Cas9 pilkkominen haluttu sekvenssi, me ilmaisi crRNA-tracrRNA fusion selostukset, jäljempänä opas RNAs (gRNAs), alkaen muutettu ihmisen H1-polymeraasi III promoottori., H1-promoottorin 3′ loppua muutettiin siten, että grnas-transkriptio voitiin aloittaa millä tahansa nukleotidilla. Rajoittaa vain vaatimus, että 20 bp crRNA kohde, jota PAM järjestyksessä, NGG (jossa N on mikä tahansa nukleotidi), tämä lähestymistapa voi periaatteessa käyttää kohdistaa mitään genomista sijainti, joka on muotoa N20NGG. Grna-ekspressiovektorin kloonauksen helpottamiseksi käytimme tyypin IIs-rajoitusentsyymiä, BsaI: ta. Tämä edellyttää synteesi 24 bp oligonukleotidi, joka sisältää alueen täydentävyyden tavoite sivuston DNA: ta., Yksinkertainen ja tehokas pöytäkirja kloonaus gRNA ilme vektori (Kuva 1A) on kuvattu yksityiskohtaisesti oheismateriaali.
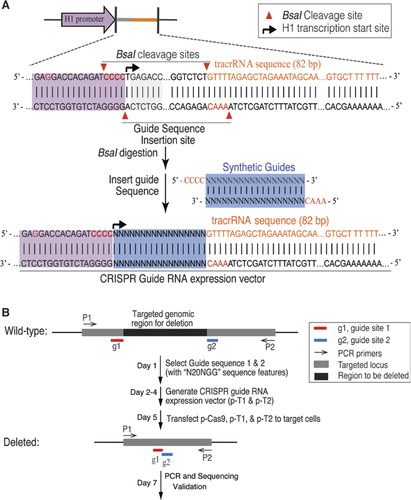
(A) ohjaus-RNA: n (gRNA) ilmaisuvektorin suunnittelu. Vektori tarkoituksena oli tuottaa gRNA selostukset synteettinen gRNA fuusioitu trans-aktivointi RNA/CRISPR RNA (tracrRNA)., H1-promoottoria muutettiin eliminoimaan sisäinen tyypin IIS-rajoitusentsyymi BsaI-sivusto vaihtamalla Gagacc H1-promoottorissa gaggacc: ksi. A BsaI-sivusto otettiin käyttöön luoda kloonaus-sivustoja gRNA ja tracrRNA fusions, joita lisätään synteettinen oligonukleotidi, duplexes kanssa yhteensopiva ulokkeita. H1-promoottorin 3′ loppua muutettiin CCACAGATCCC: ksi helpottamaan gRNAs-transkriptiota minkä tahansa nukleotidin kanssa 5′ lopussa. B) kohdennetun geenien poiston vaiheet CRISPR/Cas9: llä.,
poistamista suuri osa genomista DNA: ta, käytimme pari gRNAs vastaan suunnattu locus (Kuva 1B). Kohdealueen rajalle valittiin kaksi maalikohdetta, joissa on kuvio N20NGG. Tehokkuusetujen kohdennettuja poistetaan ohjaavat erilaisia yhdistelmiä gRNA paria määritettiin PCR-analyysia käyttäen alukkeita reunustavat kohdennettuja alueita. Villityyppiset ja typistetyt genomifragmentit ratkaistiin geelielektroforeesilla. PCR: n vahvistusviivan välttämiseksi prosentuaalinen poisto kvantifioitiin RT-PCR: llä yhdellä primer-parilla., Alukkeet oli suunniteltu poikki poistetaan junction (yksi pohjamaali ulkopuolella poistaminen, alueen, muiden primeriä poistetaan alueen) sisällä tai poistetaan alueita (sekä pohjamaalit etsi sisällä poistetaan alue). Näin ollen vain yksi bändi on täydennetty kanssa alukepari sekä tietyt solut ja hallita soluja. Laskimme prosentuaalisen poiston vertaamalla PCR-tuotteiden suhteellista määrää (kohdesolut vs. kontrollisolut), joka on vahvistettu samalla primer-parilla. Kohdennettuja poistoja tarkistettiin edelleen jaksottamalla.,
arvioida, miten gRNA paria saattaa vaikuttaa myöhemmin korjaus-ja sukupolven poistot, meidän on ensin suunniteltu sarjaa gRNAs kohdistettu ihmisen CDC42 genomisen lokuksen ja erotettu etäisyydet vaihtelevat noin 200 10 000 bp (Kuva 2A ja Täydentävä Taulukko S1). Tämän jälkeen arvioimme jokaisen gRNA-parin kykyä tuottaa poistoja ihmisen HEK 293t-soluissa Cas9: n läsnä ollessa. QPCR vahvisti nhej-pohjaisten poistojen tehokkuusedut (enintään 68 prosenttia) (Kuva 2b–2D)., Jopa poistetaan 10 kb genomista aluetta, saimme kohdistaminen hinnat 16%: sta 28%, riippuen gRNA pari. Tämä RNA-välitteinen editointi prosessi oli nopea, ja ensimmäinen havaittavissa poistetaan esiintyy noin 12 s post-transfektion (Täydentävä Kuva S1). Järjestelmä oli tehokas useissa solutyypeissä, kuten PC3 -, SK-Hep1-ja HeLa-soluissa (täydentävä Kuva S2).

(A) Kaaviokuva kuvaa sijainnit opas RNAs (gRNAs) kohdistaminen CDC42 locus. (B–D) kohdennetun poiston tehokkuus CRISPR/Cas9: llä määritettiin PCR: n avulla HEK 293t-soluissa. Alustoja käytettiin odotettujen poistoalueiden ulkopuolella. Prosenttia poistoista oli kvantifioidaan RT-PCR-alukkeita koko risteys kuluessa tai poistaminen alueella. (E–F) tehokkuus ja tarkkuus kohdennettuja poistetaan CDC42 vahvistettiin (E) Sanger sekvensointi ja (F) korkea suoritusteho sekvensointi analyysit. PCR-tuote, joka sisältää vain deleetio ampliconin, rikastettiin sekvensointia varten.,
Poistot olivat edelleen vahvisti sekvensoimalla PCR-tuotteet kattavat odotettavissa pilkkominen sivustoja. Sanger sekvensointi osoitti poistetaan liittymissä johtui siitä, että tarkka ligaatio tylppä-päättyi DSBs luoma Cas9; jokainen DSB tapahtui täsmälleen 3 bp ylävirtaan PAM järjestyksessä (Kuva 2E ja Täydentävä Kuva S3). Käytimme myös syvä sekvensointi poistetaan amplicons arvioida tarkkuutta poistetaan tehokkuutta; noin 80 prosenttia lukee, kohdennettuja DSBs olivat täysin korjata (Kuva 2F).,
– Meillä on toistanut nämä havainnot testaamalla gRNA paria tarkoituksena on poistaa katkelmia genomisen lokuksen, joka sisältää microRNA miR-21-geenin HEK 293T-soluissa. Kaksi gRNAs oli suunniteltu kohdistamaan rajat miR-21 hiusneula (täydentävä Kuva S4). Poiston tehokkuus oli 38% seuraavat transfektion kaksi gRNAs ja Cas9, mitattuna käyttäen PCR assay (Täydentävä Kuva S4B). Syvä sekvensointi vahvisti poiston tapahtuneen juuri odotetusti (lisäluku S4C).,
tutkia, onko CRISPR/ Cas9-välitteisen geenien poistaminen järjestelmä on vaikuttanut transkription valtion kohdennettuja geenit, geeni, joka koodaa kemokiinin (C-C motif) ligandin 2 (CCL2) oli kohteena. CCL2 on pieni sytokiini kuuluvat CC kemokiinin perhe, CCL2 geeni on tavoite, NF-kB signalointi. Valitsimme satunnaisesti kahdeksan kohdetta, jotka sijaitsevat ccl2-geenin lokuksen 5′ ja 3 ’ päissä (Kuva 3a). Saavutimme geenin eri alueiden vankan ja tehokkaan poistamisen käyttämällä Cas9: ää ja erilaisia gRNAs–pareja HEK 293t-soluissa (kuva 3B-3D).,

(A) Kaaviokuva kuvaa sijainnit opas RNAs (gRNAs) kohdistaminen CCL2 locus. (B–D) ccl2: n kohdennetun poistamisen tehokkuus CRISPR/Cas9: llä HEK 293t-soluissa. (E) ccl2 mRNA-arvot määritettiin tuumorinekroositekijä α: n (TNF-α) lisäyksen yhteydessä kvantitatiivisilla RT-PCR-analyyseillä HEK 293t-soluissa. Tiedot näytettiin kolmena kappaleena ± sem: llä., (F) ccl2: n kohdennetun poistamisen tehokkuus CRISPR/Cas9: llä TNF-α-hoidon jälkeen 24 tunnin ajan HEK 293T-soluissa. (G) taso luciferase toimintaa sytomegalovirus (CMV), tai SV40, tai perus järjestäjä HEK 293T-soluissa. Tiedot näytettiin kolmena kappaleena ± sem: llä. (H) Hyötysuhde kohdennettuja poistaminen luciferase geeni ohjaa CMV, tai SV40, tai perus järjestäjä HEK 293T-soluissa. (B–D,F,H) prosenttia poistaminen oli kvantifioidaan RT-PCR-alukkeita koko risteys kuluessa tai poistaminen alueella.,
Ilmaus CCL2 geeni oli dramaattisesti aiheuttama TNF-α (enintään noin 300-kertaiseksi) vuonna HEK 293T-soluissa (Kuva 3E), joka tarjoaa hyvän mallin tutkia, onko CRISPR/ Cas9-välitteisen kohdennettuja genomin muokkaus vaikuttaa transkription toimintaa. Kiinnostaa, tehokkuutta kohdennettuja poistetaan CCL2 geenin locus ei vaikuttanut hoitoon solujen TNF-α(Kuva 3F), mikä viittaa siihen, että kirjoitusasu ei muuta CRISPR/Cas9-välitteisen poistetaan., Edelleen vahvistaa tätä tulosta, me kohdennettuja eksogeeninen reportteri geeni ohjaa eri vetäjät, joilla on erilaiset vahvuudet, missä eri transkription toimintaa voitaisiin arvioida käyttäen luciferase assay (Kuva 3G). PCR-testeissä kävi ilmi, samanlainen työkalu kohdennettua poistot HEK 293T-soluissa sen jälkeen, kun co-transfektion reportteri geeni yhdessä Cas9 ja gRNA paria (Kuva 3H). Tämä tulos osoittaa, että NHEJ-välitteinen korjaus voi tapahtua huolimatta esiintymisestä eri asteista transcriptional toimintaa.,
DSBs voi edistää HDR mahdollistaa erittäin tarkka korvaaminen vaurioituneen alueen homologisia luovuttajan. Saada kohdennettuja genomista DNA: n vaihto, me esitteli pari gRNAs, Cas9, ja lineaarinen luovuttajan kanssa homologinen kohdealueen soluihin (Kuva 4A). Lineaarinen luovuttaja saatiin PCR-vahvistuksella, jossa primereillä oli 50 bp homologista sekvenssiä. Tämä sama luovuttaja lisättiin onnistuneesti ZFN-pohjaisella HDR-korjausjärjestelmällä (18)., Testata toteutettavuus CRISPR/ Cas9-välitteisen korvaaminen HDR, me kohdennettuja CCL2 locus pari gRNAs (#39 #1854 Kuvassa 3) ja luovuttajan laakeri tehostettua vihreää fluoresoivaa proteiinia (EGFP) koodaus järjestyksessä ja SV40 poly(A) – sivusto (Kuva 4A; sekvenssit ja kantoja on esitetty oheismateriaali). Käyttämällä tätä järjestelmä, noin 0,5% kohdennettuja solut olivat EGFP-positiivisia, kun taas vain 0.023% oli EGFP-positiivinen pilkata transfektion solut (vain transfektoiduissa luovuttaja), joka oli samanlainen valvonta-solut (0.021%, ilman transfektion)., Tämän jälkeen EGFP-positiiviset solut lajiteltiin virtaussytometrialla. Site-specific integration vahvistettiin PCR käyttämällä kahta paria alukkeita reunustavat sekä homologisia kädet ja koko korvannut alueella. Kuten Kuvassa 4B, havaitsimme odotettavissa korvata alueeseen, jossa täyspitkä EGFP järjestyksessä ja homologisia aseiden (Sanger sekvensointi tulos näkyy oheismateriaali). Myös endogeeninen villin tyypin alleeli havaittiin (Kuva 4B), mikä osoittaa, että kaikki alleelit eivät ole kohteena., Lisäksi valitsimme yhden klooneja EGFP-positiivisia soluja ja totesi, että kaikki kloonit (6 of 6 tutkinut) oli odotettavissa integraatio (Kuva 4C), mutta endogeenisen villi-tyyppinen alleeli oli myös havaita kolme klooneja (Kuva 4C), mikä viittaa siihen, että vain yksi alleeli, oli kohdennettava niille klooneja. Ilmaus EGFP-proteiinin kohdennettuja solut (EGFP-positiivinen lajitella soluja) oli säännelty TNF-α-hoitoa kuin arvioitiin Western blot-ja fluoresenssi-aktivoida solun lajittelu (FACS) (Kuva 4, D ja E)., Nämä tulokset osoittivat, että CRISPR/Cas9-järjestelmä voidaan luoda geeni/domain tilalle korkea hyötysuhde ja tarkkuutta.

(A) Kaavamaisen kaaviot kuvaavat menettelyn kohteena geenin korvaaminen käyttäen CRISPR/Cas9 ihmisen soluissa., Voit testata tehoa kohdennettuja geenin korvaaminen, opas RNAs (gRNAs) tarkoituksena oli poistaa merkitty alue (merkitty sivustoja 1 ja 2) CCL2 geeni ja korvata poistetut alueen EGFP-polyA kasetti luovuttajan kanssa käsivarret kanssa lyhyt alueilla homology. Kohdennettuja sivustoja, sivuston 1 ja sivuston 2 #39 #1854 sisällä CCL2 geeni on esitetty Kuviossa 3. Homologisia sekvenssejä (50 bp) ovat vain ylävirtaan ja alavirtaan poistetaan sivustoja. Asennot ja sekvenssit yksityiskohtaisesti esitetään täydentävässä materiaalissa., B) kohdennetun geenikorvauksen tehokkuus CRISPR/Cas9: llä määritettiin PCR: n avulla HEK 293t-soluissa. Pohjamaalit ulottuu liittymissä välillä CCL2 ja EGFP käytettiin PCR-monistus. C) PCR-määritys yksittäisten kloonien kohdennetusta geenikorvauksesta. (D,E) Ilmaus EGFP-proteiinin, kun TNF-α lisäksi HEK 293T-soluissa määritettiin (D) Western blot-ja (E) FACS.
Tässä me kuvata yksinkertainen ja tehokas lähestymistapa geenin poistaminen käyttämällä CRISPR/Cas9-järjestelmä., Olemme osoittaneet, että tämän järjestelmän käyttöönoton osaksi ihmisen HEK 293T-solut, ja muut ihmisen solun tyypit, aiheuttama poistot palasia jopa 10 kb hyötysuhteet vaihtelevat välillä 11% ja 68%, riippuen kohdennettuja järjestyksessä. Kyky tehokkaasti ja tarkasti poistaa genomista segmenttiä helpottaa tutkimuksen toiminnallinen genomien osia ihmisen soluissa. Tätä lähestymistapaa voidaan mahdollisesti käyttää mihin tahansa genomiseen lokukseen.
CRISPR / Cas9-järjestelmän (19-21) spesifisyys on herättänyt huolta., Sulkea pois ei-toivotut fenotyypit, koska kohde-mutaatioita, suosittelemme, että vähintään kaksi eri paria gRNAs, jota käytetään kunkin kohde-alueella. Meidän tutkimus, tarvitaan useita gRNA paria ei ole suuria rajoituksia, koska yksinkertaisuus ja erittäin tehokkuudesta tätä järjestelmää. On huomattava, että samalle alueelle suunnatut eri grnaparit toimivat tehokkaasti (Kuvat 2 ja 4). Toinen lähestymistapa ei-toivottujen mutaatioiden välttämiseksi on kaksoisnimitysmenetelmän (22, 23) käyttö., Käytimme onnistuneesti myös kaksoisnimitysmenetelmää genomisen DNA: n poistoon, mutta tehokkuus oli huomattavasti pienempi.
Se on tiedossa, että korjaus DNA DSBs on pitkälti välittyvät virhe-altis NHEJ, jossa molemmat päät ovat käsitelty ja liitettiin yhteen tavalla, joka on usein mukana nukleotidin lisäykset ja poistot. Tällainen virhealttiiden päätyliitosten havaittiin korjaamisessa DSBs luoma ZFNs tai TALENs. Sen sijaan, korjaus DSBs syntyy Cas9 ja kaksi gRNAs oli hyvin tarkka., Meidän tulokset viittaavat siihen, että tauot ovat liitettiin suoraan ilman loppua käsittely, paljastaen aiemmin arvosteta etu NHEJ polku. Tarkkoihin ligaatioihin johtava mekanismi on vielä selvittämättä. Yksi mahdollisuus on, että kohdennettu poistaminen käyttämällä Cas9 ja kaksi gRNAs johtaa risteykseen, jota kumpikaan alkuperäinen gRNAs ei tunnista. Olemme myös analysoineet tehokkuutta tuottaa indel mutaatioita yksittäisten gRNA ja gRNA pari (#39 #224 gRNA Kuva 3A), joita Sanger sekvensointi PCR amplicons (TA kloonaus)., Kiinnostaa, havaitsimme, että työkalu tuottaa indel mutaatioita yhden gRNA oli melko alhainen (9.5%, 2 21 klooneja varten #39 gRNA; 5%, 1 20 klooneja varten #224 gRNA). Kuitenkin gRNA pari syntyy korkea hyötysuhde indel mutaatioita (50%, 10, 20 klooneja varten #39 #224), joka oli samanlainen analyysi käyttämällä qPCR (52%, Kuva 3B). Ehdotamme, että yksi gRNA johtaa usein yhteen tylppään päähän katkaisukohdasta, jonka nhej korjaa tarkasti. Näin ollen mutaation tuottamisen tehokkuus on paljon pienempi yhden gRNA: n avulla kuin grnaparin.,
tunnustukset
tätä työtä tukivat kansallisen terveysinstituutin avustukset (Ei. DP1CA174421) ja W. M. Keck-säätiön C.-Z. C, ja National Natural Science Foundation of China (Nro 81101481) ja Shanghai Medical Talent koulutusohjelma (Ei. XYQ2011048) S. L. H. Tämä paperi on asetettu NIH Public Access Policy.
kilpailevat intressit
tekijät julistavat, ettei kilpailevia intressejä ole.
lisätiedot
Voit tarkastella täydentäviä tietoja, että mukana tämä paperi käy lehden verkkosivuilla osoitteessa: www.,future-science.com/doi/suppl/10.2144/000114196