Pontos gén deléció és csere a CRISPR / Cas9 rendszer segítségével az emberi sejtekben
Itt megmutatjuk, hogy két útmutató RNS párosul Cas9 hatékonyan generál DNS deléciók akár 10 kb az emberi sejtekben egy olyan folyamatban,ahol a javítás a deléció nagyrészt a pontos végcsatlakozás. Ezenkívül olyan adatokat szolgáltatunk, amelyek azt mutatják, hogy a CRISPR/Cas9 rendszer lineáris homológ javító donor jelenlétében helyettesítheti a nagy genomikus töredékeket.,
a bakteriális csoportosulás rendszeresen összefonódott rövid palindromiális ismétlődések / CRISPR-asszociált (CRISPR/Cas) loci kódolja az RNS-vezérelt immunrendszereket, amelyek védik a sejteket a behatoló vírusok és plazmidok ellen (1, 2). A Streptococcus pyogenes esetében a II-es típusú CRISPR / Cas rendszerek RNS-vezérelt endonukleázt (rgen), Cas9-et használnak a cél DNS-szekvenciák helyspecifikus hasításának katalizálására., Célzás a Cas9 konkrét genomikai oldalak által közvetített 20 nukleotid útmutató sorozat belül társult CRISPR RNS (crRNA) igényel a trans-aktiválása crRNA (tracrRNA) az újoncok a crRNA a Cas9 komplex (3). A hasítási helyek felismerését crRNA-DNS bázispárosítás és protospacer-szomszédos motívum (PAM), a DNS-komplementer régióhoz (4) illesztett három nukleotid szekvencia (NGG) határozza meg., Figyelemre méltó, hogy a tracrna-crRNA komplexet utánzó egyetlen vezető RNS (grna) képes Cas9-et felvenni a célzott genomikus helyekre, és kettős szálú töréseket (DSBs) generálni a DNS-ben (5). A CRISPR/Cas9 rendszereket különböző sejttípusokban és organizmusokban (6-12) helyspecifikus genomszerkesztésre adaptálták.
A CRISPR / Cas9 genomszerkesztését egy DSB bevezetésével indítják el egy célzott genomikus locuson az RNS-programozott RGEN segítségével. Ezt követi a DSB javítása homológia által irányított javítással (HDR) vagy nem homológ végcsatlakozással (NHEJ)., Homológ javító donor jelenlétében a CRISPR/Cas9 rendszer a HDR-eljárás révén pontos és meghatározott módosításokat és beillesztéseket hozhat létre egy célzott lokuszon. Homológ javító donor hiányában a CRISPR/Cas9 által generált egyetlen DSBs-t a hibára hajlamos NHEJ-n keresztül javítják, ami beillesztési vagy deléciós (indel) mutációkat eredményez. A kódoló exonok Indel mutációi idő előtti stop kodonokat vagy frame-shift mutációkat vezethetnek be, ezáltal inaktiválva a megfelelő fehérjéket., Az egyetlen DSB javításából származó Indel-mutációk nem feltétlenül hasznosak olyan kísérletekben, amelyek a fehérjekódoló gének funkcionális tartományainak jellemzésére vagy genomiális elemek, például intergén vagy intronikus szabályozási szekvenciák vagy nem kódoló RNS gének inaktiválására irányulnak. DNS fragmentum törlések cél loci adna egy utat, hogy tanulmányozza ezeket a funkcionális elemeket. Ebből a célból több DSB-t vezettek be, hogy deléciókat generáljanak Drosophilában (12, 13), zebrafishben (14) és emberi sejtekben (8), bár alacsony hatékonysággal., Célzott genomikus DNS-deléciókat is elértek cink ujj nukleáz (ZFN) vagy transzkripciós aktivátor-szerű effektor nukleáz (TALEN) alkalmazásával humán sejtekben (15-17). E megközelítések hatékonysága azonban általában alacsony. Emellett a ZFNs és a TALENs továbbra is nehezen és drágán tervezhető, fejleszthető és empirikusan tesztelhető a celluláris kontextusban.
itt megvizsgáltuk a CRISPR/Cas9 rendszer által katalizált emberi sejtekben a fragmentum törlések generálását. Megmutatjuk, hogy a Cas9-vel párosuló 2 grna hatékonyan képes akár 10 kb-os DNS-törléseket létrehozni., Érdekes, azt találtuk, hogy a Törlés folyamatának javítása nagyrészt a pontos végcsatlakozással történik. Ezenkívül úgy tűnik, hogy a CRISPR/Cas9 használatával történő célzott törlés független a megcélzott locus transzkripciós státusától. Végül megmutatjuk, hogy a CRISPR/ Cas9 rendszer nagy genomi töredékek helyettesítésére használható lineáris homológ javító donor jelenlétében.
anyag és módszerek
plazmid Építés
az alap H1 promoter plvthm plazmidból (Addgene, #12247, Cambridge, MA) lett erősítve., A módosított H1 promoter-t és a kívánt gRNA-szekvenciák gerincét tartalmazó oligonukleotidokat két BsaI-teleppel szintetizálták (PAN Facility, Stanford University). A kapott teljes hosszúságú termékeket PCR-vel erősítették, majd a pUC19 vektorba klónozták. Az ampicillin gén (amp) és a H1 promoter a pUC19 vektorban bsai restrikciós enzimhelyeket tartalmaz; ezeket mutálták (az amp gént g1601c-ről módosították, ami nem változtatja meg az aminosav szekvenciát; a H1 promoter gagacc-ről GAGGACC-re változott), hogy megszüntesse a bsai helyeket., A Grna klónozás protokollját a Kiegészítő anyag tartalmazza. Az összes célhelyszekvenciát az S1 kiegészítő táblázat tartalmazza.
sejttenyészet
HEK 293T, SK-Hep1 és HeLa sejteket tenyésztettek Dulbecco “s módosított Eagle” s közegben (DMEM), kiegészítve 10% magzati szarvasmarha szérummal (FBS) (Hyclone, Logan, UT) és penicillin/sztreptomicinnel (Pen/strep) (Invitrogen, Carlsbad, CA). A PC3 sejteket RPMI-1640 közegben tenyésztették, kiegészítve 10% FBS-vel és pen/strep-vel., A tumor nekrózis faktor α (TNF-α) stimulációja esetén 293t-sejtet kezeltek TNF-α(R&D Systems, Minneapolis, MN) jelzett koncentrációival. A sejteket 37°C-on és 5% CO2-on tartották párásított inkubátorban.
a célzott DNS-deléciót
HEK 293t sejteket 12-kútlemezben vetettük be, 100 000 sejt / kút sűrűségében. 24 óra elteltével a sejteket 1 µg Cas9 plazmiddal (Addgene, #41815), 0,5 µg Grna T1-gyel és 0,5 µg Grna T2 plazmiddal (Invitrogen) transzfektálták a gyártó protokolljai szerint., A genomikus DNS-t 48 órával a transzfekció után extrahálták QuickExtract DNS extrakciós oldattal (Epicentrum biotechnológiák, Madison, WI). A közös PCR-t a célzott régió erősítésére végezték a célzott régiókat szegélyező primerek segítségével. A vad típusú és csonka genomiális fragmentumokat gél-elektroforézissel oldották meg. A valós idejű PCR-t (RT-PCR) a Törlés százalékának számszerűsítésére hajtottuk végre a primerek segítségével a csomóponton vagy a törlési területen belül. Az összehasonlító Cq módszert alkalmazták a célrégió kifejezésszintjének egy referenciarégióhoz (ACTB locus) viszonyított kiszámításához., A célrégióban a deléció százalékát a célsejtek aránya a kontrollsejtekhez viszonyítva tovább számította. Az összes primer szekvenciát az S2 kiegészítő táblázat tartalmazza.
Célszekvenálás
a sejteket a transzfekció után két nappal szüretelték, és a genomikus DNS-t QuickExtract DNS extrakciós oldattal (Epicentrum biotechnológiák) extrahálták. PCR végeztek, hogy felerősítse a célzási régió genomiális DNS származó sejtek amplicons voltak mély szekvenált által MiSeq Személyes Sequencer (Illumina, San Diego, CA).,
Targeted DNA replacement
The linear donor was generated by PCR from pGl3-GFP-SV40pA plasmid, created by replacing the Renilla gene with the GFP gene in pRL-TK (Promega, Madison, WI). The primer sequences used for PCR were:
CCL2-donor-F
A*C*AGCAGCCAGAGGAACCGAGAGGCTGAGACTAACCCAGAAACATCCAATGCTTTTACGCGTCCTAGCG
CCL2-donor-R
C*A*AAAATATATTTATTTGGTGTAATAG TTACAAAATATTCATTTCCACAACCACCTGGATCCTTATCGA
The underlined regions indicate the termini of analogous oligonucleotides with 50 bp of CCL2 homology., A donor szekvenciákat a Kiegészítő anyag tartalmazza. A két 5′-legtöbb kapcsolat foszforotioát (csillaggal jelölve). A 6-kútlemezek sejtjeit 2,0 µg Cas9 plazmiddal, 0,8 µg Grna T1 plazmiddal, 0,8 µg Grna T2 plazmiddal és 0,4 µg lineáris donorral, 2000 Lipofektamin (Invitrogén) alkalmazásával transzportálták. A transzfekciót követő 48 órában a sejteket 1 ng / mL TNF-α-val kezelték 24 órán keresztül, majd GFP-pozitív sejteket válogattak.
Luciferase assay
a luciferase assay esetében a HEK 293t sejteket 96-kútlemezben vetettük be, 5000 sejt / kút sűrűségében., Után 24 h, a sejtek átmenetileg transzfektált 5 ng a pRL-TK Renilla luciferáz riporter pedig 100 ng a luciferáz riporter a cytomegalovírus (CMV), SV40 (Majom vírus 40), vagy alapvető szervező. 48 óra elteltével a luciferáz aktivitást a kettős luciferáz reporter assay rendszerrel (Promega) mértük.
Western blot
a fehérjéket nátrium—dodecil-szulfát-oldal (SDS-oldal) választotta el, és nitrocellulóz membránokra helyezte át. A membránokat 5% zsírmentes tejjel blokkolták, és GFP antitesttel inkubálták (CST, #2555S, Danvers, MA)., Az antigén-antitest komplexet fokozott kemilumineszcencia reagensekkel detektáltuk.
eredmények és megbeszélések
a II. típusú CRISPR/ Cas9 rendszert adaptáltuk a genomikus DNS mutagenizálására az emberi sejtekben. Az S. pyogenes Cas9 C-terminus SV40 nukleáris lokalizációs jelet hordozó humán kodon-optimalizált változatát egy korábban leírt rendszer (6) segítségével fejeztük ki. Annak érdekében, hogy a Cas9 hasítást a kívánt szekvenciához irányítsuk, a módosított humán h1 polimeráz III promoterből crRNA-tracrna fúziós átiratokat fejeztünk ki, a továbbiakban: guide RNS (grns)., A H1 promoter 3 ‘ végét úgy módosították, hogy lehetővé tegye a grnas bármely nukleotiddal kezdődő transzkripcióját. Csak az a követelmény korlátozza, hogy a 20 bp crRNA célt kövesse a PAM szekvencia, az NGG (ahol N bármely nukleotid), ez a megközelítés elvileg felhasználható bármely olyan genomikus hely megcélzására, amely n20ngg formában van. A gRNA expressziós vektor klónozásának megkönnyítése érdekében egy IIS típusú restrikciós enzimet, a BsaI-t használtunk. Ez szükségessé tette egy 24 bp oligonukleotid szintézisét, amely a DNS célhelyéhez komplementaritást tartalmazó régiót tartalmaz., A Grna expressziós vektor klónozásához szükséges egyszerű és hatékony protokollt (1a.ábra) részletesen ismertetjük a kiegészítő anyagban.
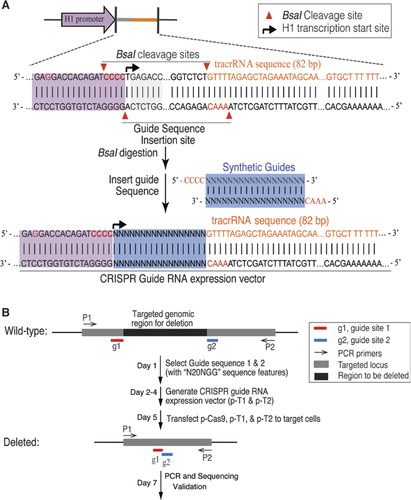
(A) a vezető RNS (gRNA) expressziós vektor kialakítása. A vektort úgy tervezték, hogy gRNA transzkripteket állítson elő egy szintetikus gRNA-val, amely a transzaktiváló RNS/CRISPR RNS-hez (tracrRNA) olvadt., A H1 promoter-t úgy módosították, hogy megszüntesse a belső IIS restrikciós enzim bsai-helyét azáltal, hogy a GAGACC-t a H1 promoter-ben GAGGACC-re változtatta. A grna és a tracrna fúziók klónozási helyeinek létrehozására bsai-telepet vezettek be szintetikus oligonukleotid-duplexek kompatibilis túlnyúlásokkal történő beillesztésével. A H1 promoter 3 ‘végét CCACAGATCCCC-re módosították, hogy megkönnyítsék a gRNAs átírását bármely nukleotiddal az 5’ végén. B) A CRISPR/Cas9 gén célzott törlésének lépései.,
a genomikus DNS nagy szegmensének törléséhez egy pár grnát használtunk a célzott locus ellen (1b ábra). A célrégió határán két, n20ngg mintával rendelkező célterületet választottak ki. A grna-párok különböző kombinációi által vezérelt célzott törlés hatékonyságát PCR-elemzések határozták meg a megcélzott régiókat szegélyező primerek segítségével. A vad típusú és csonka genomiális fragmentumokat gél-elektroforézissel oldották meg. A PCR amplifikációs torzítás elkerülése érdekében a százalékos törlést az RT-PCR számszerűsítette egy alapozó pár használatával., A primereket a deléciós csomóponton (az egyik primer a deléciós régión kívül, a másik primer a deléciós régióban) vagy a deléciós régiókon belül (mindkét primer a deléciós régióban található) tervezték. Így csak egy sávot erősítenek a primer párral mind a célzott sejtek, mind a kontroll sejtek számára. A százalékos törlést úgy számítottuk ki, hogy összehasonlítottuk az ugyanazon alapozó pár által felerősített PCR-termékek relatív mennyiségét (célsejtek kontra kontroll sejtek). A célzott törléseket tovább ellenőrizték szekvenálással.,
ahhoz, Hogy felmérje, milyen gRNA pár hatással lehet későbbi javítás, generációs törléseket, először úgy tervezték, készletek gRNAs ellen irányul az emberi CDC42 genomikai locus elválasztott távolságok kezdve mintegy 200 10.000 bp (2A Ábra-Kiegészítő Táblázat S1). Ezután megvizsgáltuk az egyes gRNA Párok azon képességét, hogy Cas9 jelenlétében törléseket generáljanak az emberi HEK 293t sejtekben. Az NHEJ-alapú törlések (legfeljebb 68%) erőteljes hatékonyságát a qPCR megerősítette (2b–2D ábra)., Még egy 10 kb-os genomikus régió törléséhez is 16% – 28% – os célzási arányt kaptunk, a gRNA pártól függően. Ez az RNS-mediált szerkesztési folyamat gyors volt, az első detektálható deléció körülbelül 12 órával a transzfekció után jelent meg (Kiegészítő S1 ábra). A rendszer számos sejttípusban hatékony volt, többek között: PC3, SK-Hep1 és HeLa sejtek (kiegészítő S2 ábra).

(A) sematikus diagram, amely a cdc42 Locust megcélzó útmutató RNS (gRNAs) helyeit ábrázolja. (B-D) A CRISPR/Cas9 célzott törlésének hatékonyságát a PCR határozta meg a HEK 293t cellákban. A várható törlési régiókon kívüli primereket használták. A Törlés százalékát az RT-PCR számszerűsítette primerek segítségével a csomóponton vagy a törlési területen belül. (E-F) A CDC42 célzott törlésének hatékonyságát és pontosságát (e) Sanger szekvenálás és (F) nagy áteresztőképességű szekvenálási elemzések igazolták. A csak a deléciós amplicont tartalmazó PCR terméket szekvenáláshoz dúsították.,
a törléseket tovább erősítették a várható hasítási helyeket lefedő PCR-termékek szekvenálásával. A Sanger-szekvenálás deléciós csomópontokat mutatott a Cas9 által létrehozott tompa végű DSB-k pontos összekapcsolásából; mindegyik DSB pontosan 3 bp-t fordult elő a PAM-szekvencia előtt (2e Ábra és S3 kiegészítő ábra). A deléciós ampliconok mély szekvenálását is használtuk a deléciós hatékonyság pontosságának felmérésére; az olvasás körülbelül 80% – ában a célzott DSB-k tökéletesen javításra kerültek (2F ábra).,
ezeket a megállapításokat a grna Párok tesztelésével állítottuk össze, amelyek célja a fragmensek törlése a mikrorna miR-21 gént tartalmazó genomikus helyből A HEK 293t sejtekben. Két grna – t terveztek a miR-21 hajtű határainak megcélzására (S4 kiegészítő ábra). A Törlés hatékonysága 38% volt a két grna-val és Cas9-vel végzett transzfekciót követően, PCR-vizsgálattal mérve (S4B kiegészítő ábra). A mély szekvenálás megerősítette, hogy a Törlés pontosan a várt módon történt (S4C kiegészítő ábra).,
annak vizsgálatára, hogy a CRISPR/ Cas9 által közvetített gén deléciós rendszert befolyásolja-e a célzott gének transzkripciós állapota, a chemokine (C-C motívum) ligand 2 (CCL2) kódoló gént célozták meg. A CCL2 egy kis citokin, amely a CC chemokine családhoz tartozik; a ccl2 gén az NF-kB jelátvitel célpontja. Véletlenszerűen nyolc célhelyet választottunk ki, amelyek a ccl2 gén locus 5′ és 3′ végében találhatók (3a ábra). A gén különböző régióinak robusztus és hatékony törlését Cas9 és különböző grna–Párok alkalmazásával értük el a HEK 293T sejtekben (3b-3D ábra).,

(A) sematikus diagram, amely a ccl2 Locust megcélzó útmutató RNS (gRNAs) helyeit ábrázolja. (B-D) A ccl2 célzott törlésének hatékonysága a CRISPR / Cas9-rel a HEK 293T cellákban. E) a ccl2 mRNS-szinteket a tumor nekrózis faktor α (TNF-α) hozzáadásával határoztuk meg kvantitatív RT-PCR elemzésekkel HEK 293t sejtekben. Az adatokat a ± sem eszközökkel három példányban mutatták be., F) a ccl2 CRISPR/Cas9-gyel történő célzott törlésének hatékonysága a TNF-α-val végzett 24 órás kezelés után a HEK 293t sejtekben. G) a cytomegalovírus (CMV) vagy az SV40, vagy a HEK 293t sejtek alapvető promoteráz aktivitásának szintje. Az adatokat a ± sem eszközökkel három példányban mutatták be. H) a CMV vagy SV40 által vezérelt luciferáz gén, vagy a HEK 293t sejtekben egy alapvető promoter célzott törlésének hatékonysága. (B–D,F,H) a Törlés százalékát az RT-PCR számszerűsítette primerek segítségével a csomóponton vagy a törlési régión belül.,
a ccl2 gén expresszióját drámaian kiváltotta a TNF-α (akár ∼300-szoros növekedés) a HEK 293t sejtekben (3e ábra), jó modellt biztosítva annak vizsgálatára, hogy a CRISPR/ Cas9 által közvetített célzott genomszerkesztést befolyásolja-e a transzkripciós aktivitás. Érdekes, hogy a ccl2 gén lokusz célzott törlésének hatékonyságát nem befolyásolta a TNF-α-val kezelt sejtek kezelése (3F ábra), ami arra utal, hogy a transzkripció nem változtatta meg a CRISPR/Cas9 által közvetített törlést., Ennek az eredménynek a további megerősítéséhez egy exogén reporter gént céloztunk meg, amelyet különböző erősségű promoterek vezetnek, ahol a különböző transzkripciós tevékenységeket luciferáz teszttel lehet értékelni (3G ábra). A PCR-vizsgálatok hasonló hatékonyságot mutattak ki a hek 293t sejtekben a reporter gén Cas9 és gRNA párokkal történő együttes transzfekciója után (3h ábra). Ez az eredmény azt jelzi, hogy az NHEJ által közvetített javítás különböző mértékű transzkripciós aktivitás előfordulása ellenére előfordulhat.,
a DSBs stimulálhatja a HDR-t, hogy lehetővé tegye a sérült régió nagyon pontos helyettesítését homológ donorral. A célzott genomikus DNS-csere érdekében bevezettünk egy Grnas-t, Cas9-et, valamint egy lineáris donort homológiával a célzott régióba a sejtekbe (4a ábra). A lineáris donort PCR-amplifikációval nyerték, 50 bp homológ szekvenciával ellátott primerekkel. Ugyanez a donor sikeresen beillesztésre került egy ZFN-alapú HDR javító rendszerrel (18)., A teszt a megvalósíthatósági a CRISPR/ Cas9 által közvetített csere által HDR, célzott az CCL2 locus egy pár gRNAs (#39 #1854 3. Ábra), valamint egy donor ellátott, megerősített zöld fluoreszcens fehérje (EGFP) kódoló szekvenciát, majd SV40 poly(A) oldal (4A Ábra; szekvenciák pozíciókat mutatja be a Kiegészítő Anyag). Ezzel a rendszerrel a célzott sejtek körülbelül 0,5%-a volt EGFP-pozitív, míg csak 0,023% volt EGFP-pozitív a mock transfection sejtekben (csak donorral transzfektálva), ami hasonló volt a kontroll sejtekhez (0,021%, transzfekció nélkül)., Az EGFP-pozitív sejteket ezután áramlási citometria szerint rendeztük. A helyspecifikus integrációt a PCR megerősítette két pár alapozó segítségével, amelyek mind a homológ karokat, mind az egész helyettesített régiót szegélyezik. A 4B ábrán látható módon megfigyeltük a teljes hosszúságú EGFP-szekvenciát és a homológ karokat (a kiegészítő anyagban látható Sanger-szekvenálási eredmény) tartalmazó várható helyettesítési régiót. Az endogén vad típusú allélt is észlelték (4b ábra), jelezve, hogy nem minden allél célzott., Továbbá, hogy a kiválasztott egyetlen klónok az EGFP-pozitív sejtek, valamint megállapította, hogy mind a klónok (6 6 vizsgálni) volt a várható integráció (4C Ábra), de az endogén vad típusú allél is kimutatható volt három klón (4C Ábra), ami arra utal, hogy csak az egyik allél volt célzott azokat a klónokat. Az EGFP-fehérje célzott sejtekben (EGFP-pozitív szortírozott sejtek) történő expresszióját a TNF-α-kezelés során a Western blot és a fluoreszcencia aktivált sejtválogatással (FAC-k) értékeltek (4., D és E ábra)., Ezek az eredmények azt mutatták, hogy a CRISPR/Cas9 rendszer nagy hatékonyságú és pontosságú gén/domain pótlások létrehozására használható.

a) vázlatos diagramok, amelyek a célzott génpótlás eljárását ábrázolják CRISPR / Cas9 alkalmazásával az emberi sejtekben., A célzott génpótlás hatékonyságának tesztelésére a guide RNS-t (grnas) úgy tervezték, hogy törölje a ccl2 gén jelzett régióját (1.és 2. hely), és hogy a törölt régiót az EGFP-polyA kazettás donorral helyettesítse a rövid homológiai régiókkal rendelkező karokkal. A 3.ábrán látható ccl2 génen belül az 1. és a 2. hely célzott helyei #39 és #1854. A homológ szekvenciák (50 bp) közvetlenül a deléciós helyek előtt és után helyezkednek el. A pozíciókat, szekvenciákat részletesen a Kiegészítő anyag tartalmazza., B) A CRISPR/Cas9-gyel történő célzott génpótlás hatékonyságát a PCR határozta meg a HEK 293t sejtekben. A PCR-erősítéshez a CCL2 és az EGFP közötti csomópontokat átívelő primereket használták. C) PCR-vizsgálat az egyes klónok célzott génpótlására. D,E) az EGFP fehérje expresszióját a TNF-α addíció során a HEK 293t sejtekben D) Western blot és E) FAC-ok határozták meg.
itt egy egyszerű és hatékony módszert írunk le a gén törlésére a CRISPR/Cas9 rendszer segítségével., Megmutattuk, hogy ennek a rendszernek a bevezetése az emberi HEK 293t sejtekbe és más emberi sejttípusokba 10 kb-ig terjedő fragmensek törlését indukálta 11% és 68% közötti hatékonysággal, a célzott szekvenciától függően. A genomiális szegmensek hatékony és pontos törlésének képessége megkönnyíti a funkcionális genomikus elemek vizsgálatát az emberi sejtekben. Ez a megközelítés potenciálisan bármilyen genomikus Loki megcélzására használható.
aggodalomra ad okot a CRISPR / Cas9 rendszer specifikussága (19-21)., A nem kívánt fenotípusok kizárása a célon kívüli mutációk miatt, javasoljuk, hogy minden egyes célrégióhoz legalább két különböző pár grna-t használjunk. Tanulmányunkban a rendszer egyszerűsége és nagy hatékonysága miatt nem volt jelentős korlátozás a több gRNA-párra való igény. Figyelemre méltó, hogy az ugyanarra a régióra irányuló különböző grna-párok nagy hatékonysággal működtek (2.és 4. ábra). A nem kívánt mutációk elkerülésének másik megközelítése a kettős nickáz módszer alkalmazása (22, 23)., Sikeresen alkalmaztuk a kettős nickáz módszert is a genomikus DNS törlésének generálására, de a hatékonyság jelentősen alacsonyabb volt.
ismeretes, hogy a DNS dsbs javítását nagyrészt a hibára hajlamos NHEJ közvetíti, amelyben a két vég feldolgozása és összekapcsolása oly módon történik, hogy gyakran nukleotid Beillesztések és törlések kísérik. Az ilyen hibára hajlamos végcsatlakozást a ZFNs vagy TALENs által létrehozott DSBs javításakor figyelték meg. Ezzel szemben a Cas9 és a két grna által generált DSB-k javítása nagyon pontos volt., Eredményeink azt sugallják, hogy a szünetek közvetlenül ligálva vannak végfeldolgozás nélkül, feltárva az NHEJ útvonal korábban nem értékelt előnyeit. Meg kell határozni azt a mechanizmust, amely a pontos ligációkat eredményezi. Az egyik lehetőség az, hogy a Cas9 és két grna-t használó célzott törlés olyan csomópontot eredményez, amelyet egyik eredeti grna sem ismer fel. Elemeztük az indel mutációk generálásának hatékonyságát az egyes GRNA-és gRNA-pároknál (#39 és #224 gRNA a 3a ábrán) a PCR-amplikonok Sanger-szekvenálásával (TA klónozás)., Érdekes, megfigyeltük, hogy az indel mutációk előállításának hatékonysága az egyetlen gRNA esetében meglehetősen alacsony volt (9,5%, 2 az 21 klónok a #39 gRNA-hoz; 5%, 1 az 20 klónok a #224 gRNA-hoz). A gRNA pár azonban az indel mutációk nagy hatékonyságát eredményezte (50%, 20 klónból 10 a #39 és #224 esetében), ami hasonló volt a qPCR alkalmazásával végzett vizsgálathoz (52%, 3b ábra). Azt javasoljuk, hogy egyetlen gRNA gyakran eredményez egy tompa végén hasítás helyén, amely pontosan javítani NHEJ. Így a mutáció generálásának hatékonysága sokkal alacsonyabb egy gRNA használatával, mint egy pár grna.,
ezt a munkát az Országos Egészségügyi Intézet (No. DP1CA174421) és a W. M. Keck Alapítvány a C.-Z. C, valamint a National Natural Science Foundation of China (no. 81101481) és Shanghai Medical Talent Training Program (No. XYQ2011048) to S. L. H. Ez a cikk a NIH nyilvános hozzáférési politikájának hatálya alá tartozik.
versengő érdekek
a szerzők nem nyilvánítanak versengő érdekeket.
kiegészítő adatok
a papírt kísérő kiegészítő adatok megtekintéséhez kérjük, látogasson el a folyóirat weboldalára: www.,future-science.com/doi/suppl/10.2144/000114196