Eliminação e substituição precisas de genes usando o sistema CRISPR / Cas9 em células humanas
Aqui mostramos que dois guias RNAs, juntamente com Cas9, geram eficientemente deleções de ADN de até 10 kb em células humanas, num processo em que a reparação da deleção é realizada em grande parte através de uma junção final precisa. Além disso, nós fornecemos dados que mostram que o sistema CRISPR/Cas9 pode substituir grandes fragmentos genômicos na presença de um doador linear de reparo homólogo.,
O bacterianas em cluster regularmente espaçadas curto palíndromo repete/ CRISPR—associado (CRISPR/Cas) loci codificam RNA-guiado sistemas imunológicos que protegem as células contra a invasão de vírus e plasmídeos (1, 2). No Streptococcus pyogenes, os sistemas CRISPR/Cas tipo II utilizam uma endonuclease guiada pelo ARN (RGEN), Cas9, para catalisar a clivagem específica do local das sequências de ADN-alvo., A focalização do Cas9 em locais genômicos específicos é mediada por uma sequência guia de 20 nucleótidos dentro de um ARN CRISPR associado (crRNA) e requer um crRNA trans-ativante (tracrRNA) que recruta o crRNA no complexo Cas9 (3). O reconhecimento de locais de clivagem é determinado por emparelhamento crRNA-DNA base e um motivo adjacente a protospacer (PAM), uma sequência de três nucleótidos (NGG) justaposta à região complementar de DNA (4)., É notável que um único Rna guia (gRNA) que imita o complexo tracrRNA-crRNA pode recrutar Cas9 para locais genômicos alvo e gerar quebras de cadeia dupla (DSBs) no DNA (5). Os sistemas CRISPR/Cas9 foram adaptados para a edição específica do genoma em diversos tipos de células e organismos (6-12).
A edição do genoma com CRISPR / Cas9 é iniciada com a introdução de um DSB em um locus genômico direcionado usando o RNA-programado RGEN. Isto é seguido pela reparação do DSB através de qualquer reparação homológica dirigida (HDR) ou não-homóloga end-joining (NHEJ)., Na presença de um dador de reparação homólogo, o sistema CRISPR/Cas9 pode ser utilizado para gerar modificações e inserções precisas e definidas num locus visado através do processo HDR. Na ausência de um dador de reparação homólogo, os DSB simples gerados pela CRISPR/Cas9 são reparados através do NHEJ propenso a erros, o que resulta em mutações de inserção ou supressão (indel). Indel mutations in coding exons may introduce premature stop codons or frame-shift mutations, thereby inactivating the corresponding proteins., Mutações Indel geradas a partir da reparação de um único DSB podem não ser úteis em experiências destinadas a caracterizar os domínios funcionais de genes codificadores de proteínas ou para a inactivação de elementos genómicos, tais como sequências regulamentares intergénicas ou intrónicas ou genes ARN não codificantes. As supressões de fragmentos de ADN no loci alvo forneceriam uma via para estudar estes elementos funcionais. Para este fim, múltiplos DSB foram introduzidos para gerar deleções em Drosophila (12, 13), zebrafish (14) e células humanas (8), embora com baixa eficiência., As deleções de DNA genômico também foram conseguidas usando nuclease de dedo de zinco (ZFN) ou nuclease de ativador de transcrição (TALEN) em células humanas (15-17). No entanto, as eficiências destas abordagens são geralmente baixas. Além disso, ZFNs e TALENs permanecem um pouco difíceis e dispendiosos de projetar, desenvolver e testar empiricamente no contexto celular.aqui, examinamos a geração de deleções de fragmentos em células humanas catalisadas pelo sistema CRISPR/Cas9. Nós mostramos que 2 gRNAs, juntamente com Cas9, podem eficientemente criar deleções de DNA de até 10 kb., De interesse, descobrimos que a reparação deste processo de exclusão é em grande parte realizada por adesão final precisa. Além disso, a supressão focalizada com CRISPR/Cas9 parece ser independente do estatuto transcritional da localidade visada. Finalmente, mostramos que o sistema CRISPR/ Cas9 pode ser usado para substituir grandes fragmentos genômicos na presença de um doador linear de reparo homólogo.
Material e Métodos
Plasmídeo construção
O básico H1 promotor foi amplificado a partir pLVTHM plasmídeo (Addgene, #12247, Cambridge, MA)., Foram sintetizados oligonucleótidos contendo o promotor H1 modificado e a espinha dorsal das sequências gRNA desejadas com dois locais BsaI (PAN Facility, Universidade de Stanford). Os produtos de comprimento completo resultantes foram amplificados pela PCR e clonados no vetor pUC19. O gene da ampicilina (amp) E o promotor H1 no vector pUC19 contêm locais enzimáticos de restrição BsaI; estes foram mutados (o gene amp foi alterado do g1601c, o que não altera a sequência de aminoácidos; o promotor H1 foi alterado de GAGACC para GAGGACC) para eliminar os locais de BsaI., O Protocolo para a clonagem de gRNA é apresentado no Material suplementar. Todas as sequências de locais de alvo são apresentadas no quadro suplementar S1.
de cultura de Células
HEK 293T, SK-Hep1, e HeLa células foram cultivadas em Dulbecco”s modified Eagle”s medium (DMEM) suplementado com 10% de soro bovino fetal (FBS) (Hyclone, Logan, UT) e penicilina/estreptomicina (caneta/strep) (Invitrogen, Carlsbad, CA). As células PC3 foram cultivadas em meio RPMI-1640 complementadas com 10% de FBS e pen/strep., Para a estimulação do factor de necrose tumoral α (TNF-α), as células 293T foram tratadas com concentrações indicadas de TNF-α(R&d Systems, Minneapolis, MN). As células foram mantidas a 37 ° C e 5% de CO2 numa incubadora humidificada.
eliminação de ADN alvo
células HEK 293T foram semeadas em placas de 12 poços com uma densidade de 100.000 células por Poço. Após 24 h, as células foram transitoriamente transfected com 1 µg de Cas9 plasmídeo (Addgene, #41815), 0,5 µg gRNA T1, e de 0,5 µg gRNA T2 plasmídeos usando Lipofectamine 2000 (Invitrogen), conforme o fabricante”s protocolos., O ADN genómico foi extraído 48 h após a transfecção utilizando uma solução de extracção rápida do ADN (epicentro Biotechnologies, Madison, WI). A PCR comum foi conduzida para ampliar a região visada usando iniciadores que flanqueiam as regiões visadas. Fragmentos genómicos de tipo selvagem e truncados foram resolvidos por electroforese gel. A PCR em tempo Real (RT-PCR) foi realizada para quantificar a percentagem de deleção usando iniciadores através da junção ou dentro da região de deleção. O método CQ comparativo foi utilizado para calcular o nível de expressão da região-alvo em relação a uma região de referência (ACTB locus)., A percentagem de eliminação na região-alvo foi ainda calculada pela razão das células-alvo em relação às células de controlo. Todas as sequências de iniciação estão listadas no quadro suplementar S2.
sequenciação-alvo
as células foram colhidas dois dias após a transfecção, e o ADN genómico foi extraído utilizando uma solução de extracção de ADN de extracção rápida (Biotecnologias epicentro). A PCR foi conduzida para amplificar a região alvo com DNA genômico derivado das células, e os amplificadores foram profundamente sequenciados pelo sequenciador pessoal MiSeq (Illumina, San Diego, CA).,
Targeted DNA replacement
The linear donor was generated by PCR from pGl3-GFP-SV40pA plasmid, created by replacing the Renilla gene with the GFP gene in pRL-TK (Promega, Madison, WI). The primer sequences used for PCR were:
CCL2-donor-F
A*C*AGCAGCCAGAGGAACCGAGAGGCTGAGACTAACCCAGAAACATCCAATGCTTTTACGCGTCCTAGCG
CCL2-donor-R
C*A*AAAATATATTTATTTGGTGTAATAG TTACAAAATATTCATTTCCACAACCACCTGGATCCTTATCGA
The underlined regions indicate the termini of analogous oligonucleotides with 50 bp of CCL2 homology., As sequências do dador são apresentadas no Material suplementar. Os dois 5′-a maioria das ligações são fosforotioato (indicado por asteriscos). Células com 6 placas foram transitoriamente transfected com 2,0 µg Cas9 plasmídeo, de 0,8 µg gRNA T1 plasmídeo, de 0,8 µg gRNA T2 plasmídeo, e 0,4 µg linear doador usando Lipofectamine 2000 (Invitrogen). Às 48 horas após a transfecção, as células foram tratadas com 1 ng/mL de TNF-α durante 24 h e, em seguida, as células GFP positivas foram ordenadas.
ensaio de Luciferase
para o ensaio de luciferase, as células de HEK 293T foram semeadas em placas de 96 alvéolos com uma densidade de 5000 células por alvéolo., Após 24 h, as células foram transfectadas transitoriamente com 5 ng de pRL-TK Renilla luciferase reporter e 100 ng de luciferase reporter com citomegalovírus (CMV), SV40 (vírus símio 40) ou promotor básico. Após 48 horas, a actividade da luciferase foi determinada com o sistema de doseamento da dupla luciferase reporter (Promega).
Western blot
As proteínas foram separadas pelo sulfato de sódio—PAGE (SDS-PAGE) e transferidas para membranas nitrocelulosas. As membranas foram bloqueadas com 5% de leite magro e incubadas com anticorpos GFP (CST, #2555S, Danvers, MA)., O complexo antigénio-anticorpo foi detectado com reagentes quimioluminescência melhorados.
resultados e discussão
adaptámos o sistema bacteriano tipo II CRISPR / Cas9 para mutagenizar o ADN genómico nas células humanas. A versão otimizada do codon humano da proteína Cas9 de S. pyogenes portando um sinal de localização nuclear SV40 C-terminus foi expressa usando um sistema anteriormente descrito (6). Para direcionar a clivagem Cas9 para a sequência desejada, expressamos transcrições de fusão crRNA-tracrRNA, a seguir denominadas guide RNAs( gRNAs), de um promotor de polimerase III humano H1 modificado., O fim de 3′ do promotor H1 foi modificado para permitir a transcrição de gRNAs que começam com qualquer nucleótido. Limitada apenas pelo requisito de que o alvo crRNA de 20 bp seja seguido pela sequência PAM, NGG (onde N é qualquer nucleótido), esta abordagem pode, em princípio, ser usada para atingir qualquer localização genómica que tenha a forma N20NGG. Para facilitar a clonagem do vetor de expressão gRNA, usamos uma enzima de restrição Tipo IIs, BsaI. Isto exigiu a síntese de um oligonucleótido de 24 bp contendo uma região de complementaridade com o local-alvo no ADN., O protocolo simples e eficiente para a clonagem do vetor de expressão gRNA (figura 1A) é descrito em detalhes no Material suplementar.
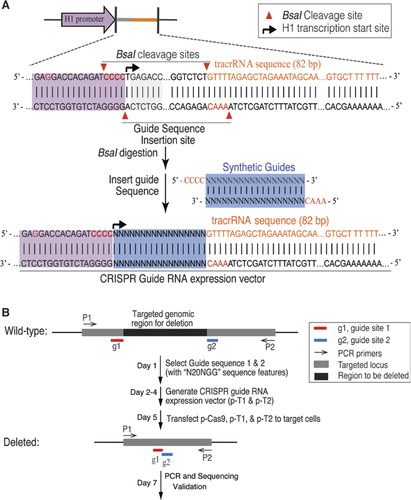
(A) Design of the guide RNA (gRNA) expression vector. O vetor foi projetado para produzir transcrições gRNA com um gRNA sintético fundido ao RNA/RNA CRISPR (tracrRNA)., O promotor H1 foi modificado para eliminar o local interno da enzima bsai de restrição do tipo IIS, alterando o gagacc do promotor H1 para o GAGGACC. Um site BsaI foi introduzido para criar locais de clonagem para perfusões de gRNA e tracrRNA por inserção de oligonucleótidos sintéticos duplexes com dentes-de-fora compatíveis. A extremidade 3′ do promotor H1 foi modificada para ser CCACAGATCCCC para facilitar a transcrição de gRNAs com qualquer nucleótido na extremidade 5′. B) as etapas da eliminação genética específica com CRISPR/Cas9.,
para a eliminação de um grande segmento de ADN genómico, usamos um par de gRNAs contra o locus-alvo (figura 1B). Dois locais alvo com o padrão N20NGG foram selecionados no limite da região alvo. As eficiências da eliminação orientada por várias combinações de pares gRNA foram determinadas por análises PCR utilizando iniciadores que flanqueiam as regiões visadas. Fragmentos genómicos de tipo selvagem e truncados foram resolvidos por electroforese gel. Para evitar o viés de amplificação PCR, a eliminação por cento foi quantificada pela RT-PCR usando um par de iniciadores., Os primers foram projetados através da junção de deleção (um primer fora da região de deleção, o outro primer dentro da região de deleção) ou dentro das regiões de deleção (ambos os primers localizam dentro da região de deleção). Assim, apenas uma única banda é amplificada com o par de iniciadores tanto para as células alvo e células de controle. Calculámos a percentagem de eliminação comparando a quantidade relativa de produtos PCR (células alvo versus células de controlo) amplificados pelo mesmo par de iniciadores. As supressões direcionadas foram verificadas através de sequenciação.,
para avaliar como os pares gRNA podem afetar a reparação e geração subsequentes de supressões, nós primeiro projetamos conjuntos de gRNAs voltados contra o loco genômico CDC42 humano e separados por distâncias que variam de aproximadamente 200 a 10.000 PB (figura 2A e tabela suplementar S1). Em seguida, avaliamos a capacidade de cada par gRNA para gerar deleções em células HEK 293T humanas na presença de Cas9. A eficácia robusta das supressões baseadas no NHEJ (até 68%) foi confirmada pela qPCR (figura 2B-2D)., Mesmo para a exclusão de uma região genômica de 10 kb, obtivemos taxas de alvo de 16% a 28%, dependendo do par gRNA. Este processo de edição mediado pelo ARN foi rápido, com a primeira supressão detectável aparecendo aproximadamente 12 h após a transfecção (figura suplementar S1). O sistema foi eficaz em uma variedade de tipos de células, incluindo: células PC3, SK-Hep1 e hela (figura suplementar S2).

(A) diagrama esquemático que representa as localizações do Guia RNAs (gRNAs) direccionado para o cdc42 locus. B-D) A eficiência da eliminação orientada com CRISPR/Cas9 foi determinada pela PCR nas células HEK 293T. Foram utilizados iniciadores fora das regiões de eliminação esperadas. A porcentagem de deleção foi quantificada por RT-PCR usando iniciadores através da junção ou dentro da região de deleção. (E-F) a eficiência e precisão da eliminação orientada em CDC42 foi confirmada por (e) sequenciação de Sanger e (F) análises de sequenciamento de alto rendimento. O produto PCR contendo apenas o fragmento amplificado de deleção foi enriquecido para sequenciação.,
as supressões foram confirmadas através da sequenciação de produtos PCR que abrangem os locais de clivagem esperados. A sequenciação de Sanger mostrou junções de eliminação resultado da ligação precisa dos Dsb de ponta romba criados pelo Cas9; cada DSB ocorreu exatamente 3 bp a montante da sequência PAM (figura 2E e figura suplementar S3). Nós também usamos seqüenciamento profundo de amplicons de exclusão para avaliar a precisão da eficiência de exclusão; em cerca de 80% das leituras, O DSB alvo foram perfeitamente reparados (figura 2F).,recapitulámos estes resultados testando pares gRNA concebidos para eliminar fragmentos de um locus genómico contendo o gene mir-21 da microRNA nas células HEK 293T. Dois gRNAs foram projetados para atingir os limites do gancho de cabelo miR-21 (figura suplementar S4). A eficiência de eliminação foi de 38% após a transfecção com os dois gRNAs e Cas9, medida utilizando um ensaio PCR (figura suplementar S4B). Sequenciamento profundo confirmou que a eliminação ocorreu exatamente como esperado (figura suplementar S4C).,
para investigar se o sistema de deleção do gene mediado CRISPR / Cas9 é influenciado pelo Estado transcritional dos genes visados, o gene que codifica o ligando quimiocina (C-C motif) 2 (CCL2) foi alvo. CCL2 é uma pequena citocina pertencente à família CC chemokine; o gene CCL2 é um alvo de sinalização NF-kB. Selecionamos aleatoriamente oito locais-alvo localizados nas extremidades de 5′ e 3′ do gene CCL2 locus (figura 3A). Conseguimos a eliminação robusta e eficiente de diferentes regiões do gene usando Cas9 e diferentes pares de gRNAs em células HEK 293T (figura 3B–3D).,
(A) diagrama esquemático representando as localizações do Guia RNAs (gRNAs) visando o CCL2 locus. B-D) A eficiência da eliminação direccionada de CCL2 com CRISPR/Cas9 em células HEK 293T. E) os níveis de ARNm CCL2 foram determinados com base na adição do factor de necrose tumoral α (TNF-α) Por análises quantitativas de RT-PCR em células HEK 293T. Os dados foram apresentados com as médias ± sem em triplicado., F) a eficiência da eliminação direccionada de CCL2 com CRISPR/Cas9 após tratamento com TNF-α durante 24 h em células HEK 293T. G) o nível de actividade da luciferase por citomegalovírus (CMV) ou SV40, ou um promotor básico em células HEK 293T. Os dados foram apresentados com as médias ± sem em triplicado. H) eficiência da eliminação direccionada de um gene de luciferase controlado por CMV, ou SV40, ou de um promotor básico em células HEK 293T. (B-D, F, H) A percentagem de deleção foi quantificada por RT-PCR usando iniciadores através da junção ou dentro da região de deleção.,
a Expressão da CCL2 gene foi drasticamente induzida por TNF-α (até ∼300 vezes de aumento) em HEK 293T células (Figura 3E), proporcionando um bom modelo para investigar se o CRISPR/ Cas9 mediada alvo do genoma de edição é afetado pela atividade transcricional. De interesse, a eficiência da eliminação focalizada do locus do gene CCL2 não foi afetada pelo tratamento de células com TNF-α(figura 3F), sugerindo que a transcrição não alterou a deleção mediada por CRISPR/Cas9., Para confirmar ainda mais este resultado, visamos um gene repórter exógeno impulsionado por vários promotores com diferentes dosagens, onde as diferentes actividades transcritionais poderiam ser avaliadas utilizando um teste de luciferase (figura 3G). Os ensaios de PCR revelaram uma eficiência semelhante em supressões direcionadas em células HEK 293T após a co-transfecção do gene repórter, juntamente com pares Cas9 e gRNA (figura 3H). Este resultado indica que a reparação mediada por NHEJ pode ocorrer apesar da ocorrência de vários graus de actividade transcriptional.,
DSBs pode estimular HDR para permitir a substituição altamente precisa da região danificada por um doador homólogo. Para obter a substituição de DNA genômico direcionado, introduzimos um par de gRNAs, Cas9, e um doador linear com homologia para a região alvo em células (figura 4A). O dador linear foi obtido por amplificação PCR com iniciadores com uma sequência homóloga de 50 bp. Este mesmo doador foi inserido com sucesso usando um sistema de reparo HDR baseado em ZFN (18)., Para testar a viabilidade de CRISPR/ Cas9 mediada substituição por HDR, que tivemos o CCL2 locus com um par de gRNAs (#39 #1854 mostrado na Figura 3) e um doador de rolamentos reforçada a proteína verde fluorescente (EGFP) sequência codificante e SV40 poli(Um) site (Figura 4A; sequências e as posições são apresentadas no Material Suplementar). Usando este sistema, aproximadamente 0.5% das células alvo eram EGFP-positivo, enquanto apenas 0.023% eram EGFP-positivo em células de transfecção mock (apenas transfected com doador), que era semelhante às células de controle (0.021%, sem transfecção)., As células EGFP-positivas foram então ordenadas por citometria de fluxo. A integração específica do local foi confirmada pela PCR usando dois pares de iniciadores flanqueando ambos os braços homólogos e toda a região substituída. Como mostrado na figura 4B, observamos a região substituída esperada contendo a sequência EGFP de comprimento completo e os braços homólogos (resultado da sequenciação de Sanger mostrado no Material suplementar). Foi igualmente detectado o alelo endógeno de tipo selvagem (figura 4B), indicando que nem todos os alelos são visados., Além disso, nós selecionamos o único clones de EGFP-células positivas e descobriu que todos os clones (6 de 6 examinado) tinha esperado integração (Figura 4C), mas o endógenos do tipo selvagem alelo foi também detectado em três dos clones (Figura 4C), sugerindo que apenas um alelo foi alvo os clones. A expressão da proteína EGFP em células alvo (células seleccionadas EGFP positivas) foi regulada para cima com o tratamento TNF-α avaliado por Western blot e fluorescence activated cell sorting (FACS) (Figura 4, D E E)., Estes resultados demonstraram que o sistema CRISPR/Cas9 pode ser utilizado para criar substituições de genes/domínios com elevada eficiência e precisão.

(A) diagramas esquemáticos representando o procedimento de substituição de genes alvo usando CRISPR/Cas9 em células humanas., Para testar a eficácia da substituição de genes alvo, o guia RNAs (gRNAs) foi concebido para eliminar a região indicada (marcada pelos locais 1 e 2) do gene CCL2 e para substituir a região eliminada pelo dador de cassetes EGFP-polyA por braços com regiões curtas de homologia. Os sites alvo do site 1 e do site 2 são # 39 e # 1854 dentro do gene CCL2 mostrado na Figura 3. As sequências homólogas (50 bp) são apenas a montante e a jusante dos locais de eliminação. As posições e sequências em detalhe são apresentadas no Material suplementar., B) a eficiência da substituição genética orientada com CRISPR/Cas9 foi determinada pela PCR nas células HEK 293T. Os iniciadores que abrangem as junções entre CCL2 e EGFP foram usados para amplificação PCR. C) doseamento de PCR para a substituição genética específica dos clones isolados. (D,E) a expressão da proteína EGFP sobre a adição de TNF-α EM células HEK 293T foi determinada por (D) Western blot e (e) FACS.
Aqui descrevemos uma abordagem simples e eficiente para a eliminação de genes usando o sistema CRISPR / Cas9., Nós demonstramos que a introdução deste sistema em células humanas HEK 293T, e outros tipos de células humanas, induziu deleções de fragmentos até 10 kb com eficiências variando entre 11% e 68%, dependendo da sequência alvo. A capacidade de eliminar de forma eficiente e precisa os segmentos genômicos facilitará o estudo dos elementos genômicos funcionais nas células humanas. Esta abordagem pode ser potencialmente utilizada para atingir qualquer loci genómico.a especificidade do sistema CRISPR / Cas9 tem suscitado preocupações (19-21)., Para descartar fenótipos indesejados devido a mutações fora do alvo, sugerimos que pelo menos dois pares diferentes de gRNAs sejam usados para cada região alvo. Em nosso estudo, a necessidade de múltiplos pares gRNA não foi uma limitação importante, dada a simplicidade e alta eficiência deste sistema. É notável que diferentes pares de gRNAs direcionados para a mesma região trabalharam com alta eficiência (Figuras 2 e 4). Outra abordagem para evitar mutações indesejadas é o uso do método da nickase dupla (22, 23)., Nós também aplicamos com sucesso o método da dupla nickase para gerar a exclusão do DNA genômico, mas a eficiência foi consideravelmente menor.
sabe-se que a reparação do ADN DSBs é largamente mediada pelo NHEJ propenso a erros, no qual as duas extremidades são processadas e ligadas entre si de uma forma que é frequentemente acompanhada por inserções e supressões de nucleótidos. Tal junção final propensa a erros foi observada no reparo de DSBs criado por ZFNs ou TALENs. Em contrapartida, a reparação da DSB gerada pelo Cas9 e por duas gRNAs foi muito precisa., Nossos resultados sugerem que as quebras são diretamente ligadas sem processamento final, revelando uma vantagem anteriormente não valorizada da via NHEJ. O mecanismo que resulta nas ligações precisas permanece por determinar. Uma possibilidade é que a exclusão orientada usando Cas9 e dois gRNAs resulta em uma junção que não é reconhecida por nenhum dos gRNAs originais. Também analisamos a eficiência na geração de mutações indel para o par gRNA e gRNA individual (#39 e # 224 gRNA na figura 3A) pela Sequenciação Sanger de amplicons PCR (Clonagem TA)., De interesse, observamos que a eficiência de gerar mutações indel para o gRNA único foi bastante baixa (9,5%, 2 de 21 clones para o #39 gRNA; 5%, 1 de 20 clones para o #224 gRNA). No entanto, o par gRNA gerou alta eficiência de mutações indel (50%, 10 de 20 clones para #39 e #224), que foi semelhante ao ensaio utilizando qPCR (52%, figura 3B). Propomos que um único gRNA muitas vezes resulta em uma extremidade contundente do local de clivagem, que será precisamente reparado por NHEJ. Assim, a eficiência de gerar mutação é muito menor com o uso de um único gRNA do que um par de gRNAs.,
agradecimentos
Este trabalho foi apoiado por subsídios do Instituto Nacional de Saúde (No. DP1CA174421) and the W. M. Keck foundation to C.-Z. C, and National Natural Science Foundation of China (No. 81101481) and Shanghai Medical Talent Training Program (No. XYQ2011048) para a S. L. H. Este artigo está sujeito à Política de acesso público da NIH.
interesses concorrentes
os autores não declaram interesses concorrentes.
dados suplementares
para ver os dados suplementares que acompanham este artigo, visite o sítio web do jornal em: www.,future-science.com/doi/suppl/10.2144/000114196